白菜官网张鹏团队在脊椎动物通用环境DNA metabarcoding检测技术上取得新进展
脊椎动物作为自然生态系统中不可或缺的生物类群,在生态系统中起着重要的作用。而近40年的监控数据表明,全球脊椎动物种群的平均数量降低了68%,对生态系统造成深远影响。为及时掌握脊椎动物的数量变化、积极应对这些变化带来的各种挑战,开展大规模生物调查并且准确监测生态系统中脊椎动物多样性的组成变动是十分必要的。然而,传统的走样线、安置地笼或红外相机等调查方法不仅需要大量的人力物力,还需要专业的分类学知识,给大规模开展脊椎动物物种调查与监测带来了挑战。
近年来,一种调查和监测生态系统中生物多样性的新兴技术——环境DNA 宏条形码 (environmental DNA metabarcoding) 技术,为大规模开展生物调查与监测带来新机遇。该方法仅需对从水、土壤或空气等环境样品中获得的DNA进行实验分析,便可获得大量在相关区域活动的生物的信息,是一种非损伤性的、灵敏度高且简单高效的了解生物多样性的分子工具。然而,该方法的准确性在很大程度上依赖于扩增特定目标序列的PCR引物。对于脊椎动物而言,目前可用的大多数引物都是为特定类群设计的,如鱼类、两栖类、哺乳类等,而适用于所有脊椎动物的“通用引物”十分有限。此外,目前的脊椎动物环境DNA调查多采用单一标记片段,物种的检出率一般为90%左右,无法可靠获取某一生态系统中全面完整的脊椎动物生物多样性信息。
为了给依赖于环境DNA metabarcoding技术开展的脊椎动物调查提供新工具,并解决单一标记片段的局限性,白菜官网张鹏教授团队通过分析NCBI数据库中约6500条脊椎动物完整的线粒体基因组数据,针对脊椎动物线粒体的12S、16S和COI三个基因,开发了一套新的脊椎动物环境DNA通用扩增引物集 (V12S-U、V16S-U和VCOI-U)。该引物集适用于所有的脊椎动物类群,且扩增子长度为200-250 bp,便于利用短读长测序平台获取数据。研究通过in silico PCR、mock DNA(混合已知物种DNA的模拟DNA)和动物园环境DNA的实验测试,评估并比较了新开发的引物与之前发表的7个引物的物种检测能力。研究结果显示,在mock DNA测试中,运用新的引物集在不同的模拟条件下均可以检测出全部的物种,效果优于其他引物(图1);在动物园环境DNA扩增测试中,新的引物集可以检测出采样园区中饲养的全部脊椎动物,且扩增结果特异性高于其他引物(图2)。理论上,同时应用新开发的3个标记片段进行环境DNA分析,脊椎动物物种的检出率可以达到99.5%。
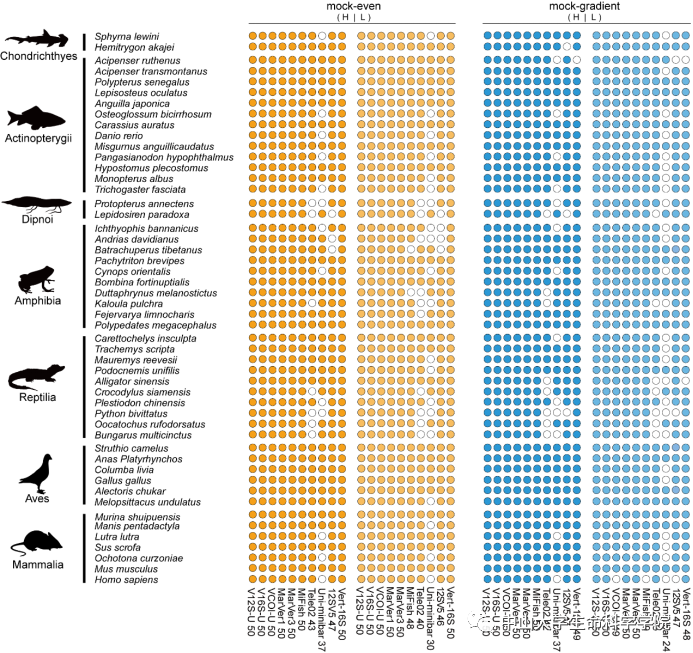
图1 新引物与前人引物的mock DNA实验对比图,
橙色为等量混合的mock DNA,蓝色为梯度混合的mock DNA,每组前三列为新开发的引物。
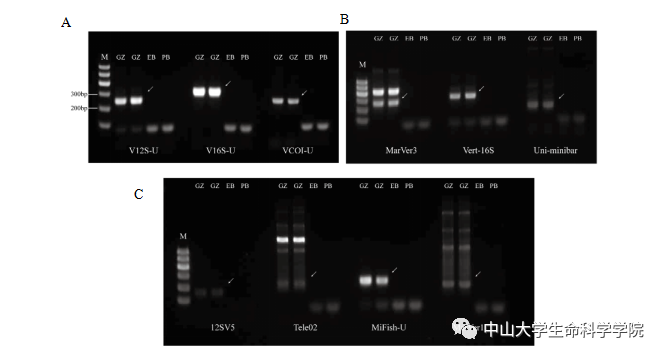
图2 新引物与前人引物对动物园环境DNA扩增特异性对比图,
A为新引物扩增结果,B、C为前人引物扩增结果。
为了进一步探究基于多标记片段的环境DNA metabarcoding技术对脊椎动物多样性的调查能力,研究团队对广东省汕尾市新田镇与河口镇进行了野外环境DNA的采集,并与同院的范朋飞教授团队合作在唐家河国家自然保护区中采集了春秋两个季度的环境DNA样品(图3)。对这些环境样品使用新开发的引物集进行脊椎动物多样性调查共检测到了895个脊椎动物的OTUs,包括182个种、195个属和94个科,其中84%的物种为存在记录的本地物种,这展现出新引物集强大的物种检测能力与通用性。同时,研究结果表明使用多个标记片段比单一标记片段可以有效提高物种的检测效率,实现更好更全面的生物多样性调查,这对于监测生态系统中的脊椎动物等大型生物尤为重要(图4)。
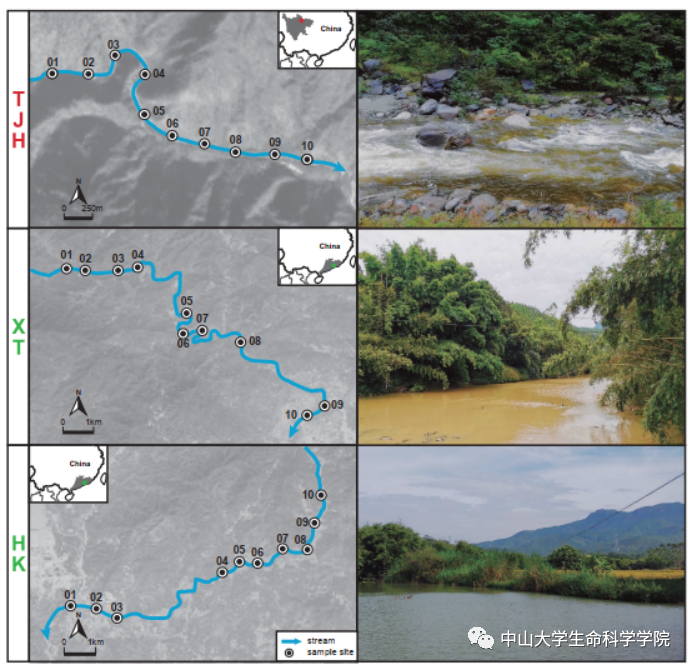
图3 野外环境DNA采样地图及实景
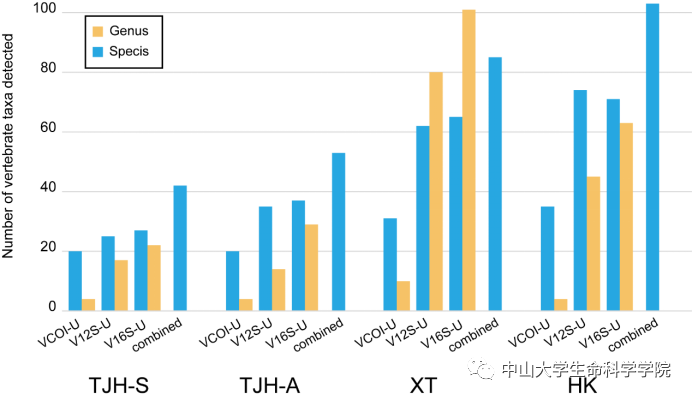
图4 使用多标记片段进行脊椎动物调查的效果
该工作为应用环境DNA metabarcoding检测技术准确全面地调查脊椎动物多样性提供了可靠的技术路线,新开发的引物集使研究人员能够在一次环境DNA metabarcoding调查中得到该区域所有脊椎动物物种的信息,大大简化了工作流程、降低了调查成本,在未来的脊椎动物生物调查中具有广泛的应用前景。
近日,该成果以“VertU: universal multilocus primer sets for eDNA metabarcoding of vertebrate diversity, evaluated by both artificial and natural cases”为题发表在国际生态学期刊Frontiers in Ecology and Evolution上,2023白菜网址官网大全为第一作者单位。本研究得到了国家自然科学基金面上项目的资助。
文章链接:
https://doi.org/10.3389/fevo.2023.1164206
作者
本院
来源
本院



