白菜官网张鹏团队在基于自制探针获取系统发育基因组学数据的方法学研究中取得新进展
重建快速分化类群的演化历史一直都是系统发育基因组学研究所面临的挑战之一。近年来的研究发现,解决这类挑战不仅需要分析基因组级别的数据,还需要同时分析不同类型的数据,如编码数据和非编码数据。目标序列捕获技术是收集上述数据的有效策略。目前大多数的序列捕获方法均是使用定制的探针集,这需要研究人员根据目标类群的基因组序列设计探针,随后由公司合成这些探针。然而,定制合成一套探针集的整个过程既耗时(至少6周)又昂贵(每个捕获反应花费约1300元人民币),更重要的是,许多非模式生物类群缺少可用的基因组资源来设计探针。
对此,张鹏教授团队开发了一种基于自制全长cDNA探针的序列捕获方法 (Full length cDNA capture; FLc-Capture) 来同时收集编码和非编码的系统发育基因组学数据(实验流程见图1,数据分析流程见图2)。FLc-Capture方法只需要研究者从目标类群中挑选一个常见物种作为探针制备物种,并从中提取高质量的mRNA,然后以此为模板合成生物素化的全长cDNA作为捕获探针。这种探针制备策略直接跳过了探针设计与合成的步骤,使研究人员可以在不需要基因组数据和商业探针合成的情况下,为任意一类生物制备一套捕获探针集,具有高度的灵活性,且特别适合非模式生物的研究。利用该方法制备一套全长cDNA探针的总时长不超过3天,每个捕获反应的花费不足10元,这比定制合成一套探针集更经济、省时。为了证明FLc-Capture方法的实用性,研究团队利用榕蛇的mRNA制备了全长cDNA探针,然后利用这些探针成功地从24个游蛇物种和12个远缘蛇类样本中捕获到数千条编码和非编码序列。所有测试样本的平均捕获效率可达到35%,不亚于商业合成探针的捕获效果。基于这些捕获数据,研究者构建了两个系统发育基因组学数据集来研究游蛇科的系统发育关系,其中数据集1包含了1,075个编码基因位点(总长度~817,000 bp),数据集2包含了1,948个非编码基因位点(总长度~111,400 bp)。最终这两个数据集推断出了高度相似且解析度良好的游蛇科系统发育树,其中有85%的节点具有>95%的自展值。上述实验测试结果表明,FLc-Capture是一种灵活、快速、经济的目标序列捕获方法,它可以同时收集编码和非编码的系统基因组学数据集,特别有助于研究生命之树中的困难进化问题。
近日,该成果以“Simultaneously Collecting Coding and Noncoding Phylogenomic Data Using Homemade Full-length cDNA Probes, Tested by Resolving the High-level Relationships of Colubridae ”为题发表在学术期刊Frontiers in Ecology and Evolution上。张鹏教授为该项工作的通讯作者,李佳璇博士后为该文第一作者。该研究得到了国家自然科学基金、中组部“青年拔尖人才计划”等项目资助。
张鹏教授团队近年来在基于自制探针获取分子系统学数据的方法学研究上佳绩不断,除了FLc-Capture方法外,该团队还开发了以PCR技术为核心的扩增子捕获技术[1-3]和适用于解析低阶元系统发育关系的AFLP基因组捕获技术[4-5]等。这些数据收集策略的上手难度并不高,一般的分子生物学实验室都可以尝试,一旦掌握这些技术后,实验室便能够自主性、规模性地开展相关的分子系统学研究。
原文链接:https://www.frontiersin.org/articles/10.3389/fevo.2022.969581/full?&utm_source=Email_to_authors_&utm_medium=Email&utm_content=T1_11.5e1_author&utm_campaign=Email_publication&field=&journalName=Frontiers_in_Ecology_and_Evolution&id=969581
想要了解更多的关于张鹏团队的科研成果,请浏览实验室网站:https://lab.rjmart.cn/#/10163/elab
相关文献:
- Zhang, Y., Deng, S. H., Liang, D., and Zhang P. (2019). Sequence capture across large phylogenetic scales by using pooled PCR–generated baits: A case study of Lepidoptera. Mol Ecol Resour. 19, 1037–1051. doi:10.1111/1755-0998.13026
- 张圆,梁丹,张鹏. 基于混合PCR产物制备捕获探针的基因组捕获方法. Bio-protocol. 2021. doi: 10.21769/BioProtoc.1010599
- Gu H. M., Zhang P., Xu M. H., and Liang D. Amplicon genome fishing (AGF): a rapid and efficient method for sequencing target cis-regulatory regions in nonmodel organisms. Molecular Genetics and Genomics. 296, 527–539. doi:10.1007/s00438-021-01775-0
- Li, J. X., Zeng, Z. C., Wang, Y. Y., Liang, D., and Zhang, P. (2019). Sequence capture using AFLP-generated baits: A cost-effective method for high-throughput phylogenetic and phylogeographic analysis. Ecol. Evol. 9, 5925–5937. doi:10.1002/ece3.5176
- 李佳璇,梁丹,张鹏. AFLP基因组捕获技术: 一种通用的低阶元的系统发育分析方法. Bio-protocol. 2021. doi:10.21769/BioProtoc.1010597
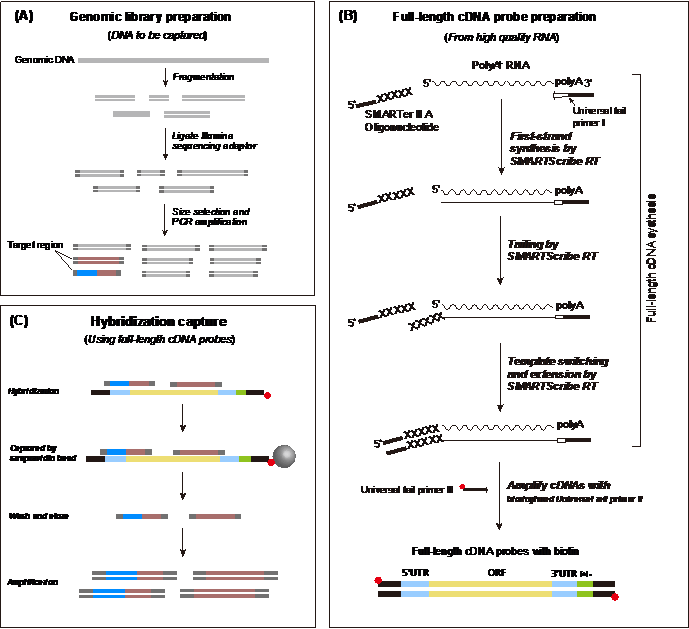
图1:FLc-Capture的实验流程
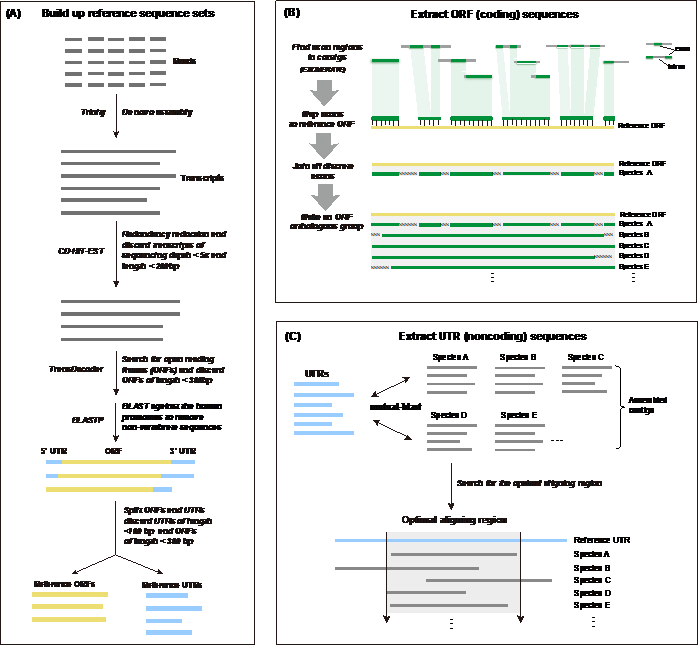
图2:FLc-Capture的数据分析流程
作者
本院
来源
本院



